Im Jahr 2019 begann SVA zusammen mit ihrem Kunden Berlin Institute of Health (BIH) und dem Bereich Forschung & Lehre im Geschäftsbereich IT der Charité mit der Ausarbeitung und Planung eines Data Lakes für die medizinische Forschung. Im Rahmen von Data Value Workshops wurden zusammen mit dem Kunden die vorhandenen Problemstellungen analysiert, Datenquellen betrachtet und eine langfristig tragfähige Lösung erarbeitet.
Definition des übergeordneten Ziels
Das große Ziel bestand darin, eine zentrale Data-Lake-Plattform zu schaffen. Dabei galt es strukturierte und unstrukturierte Forschungs- sowie klinische Daten zentral mit einem einheitlichen Sicherheits- und Berechtigungskonzept zur Verfügung zu stellen und zu integrieren.
Der Data Lake konnte noch im selben Jahr in einen Pilotbetrieb genommen werden und wird inzwischen bereits von vielen Datenquellen gespeist. Ein Team von SVA Experten arbeitet zusammen mit den Experten von BIH und der Charité an der Bewirtschaftung des Data Lakes und an der Erstellung von neuen Data Assets sowie der Umsetzung von unterschiedlichen Use Cases.
Außerdem ist dieser ein maßgeblicher Teil der Health Data Plattform (HDP) und damit ein wesentlicher und integraler Bestandteil bei der Digitalisierungsstrategie der Charité. Die nun einfachere und schnellere Nutzung von Forschungs-und Gesundheitsdaten führt schon jetzt zu einer stärkeren Vernetzung der Forschungseinrichtungen und bringt neue Impulse für den Bereich digitale Medizin.
Aufgabe
Im Rahmen der SARS-CoV2-Pandemie konnte der Data Lake sein Potential aufzeigen. Die Aufgabe bestand darin, einen tagesaktuellen Überblick über die Krankheitsverläufe der Patienten zu visualisieren, die an COVID-19 erkrankt sind und sich im Verlaufe des Krankenhausaufenthalts auf der Intensivstation befinden. Der Gesundheitszustand und der Schweregrad der Krankheitsentwicklung sollte dabei über die Zeit mit Hilfe des SOFA-Score[1] dargestellt werden.
Technische Umsetzung
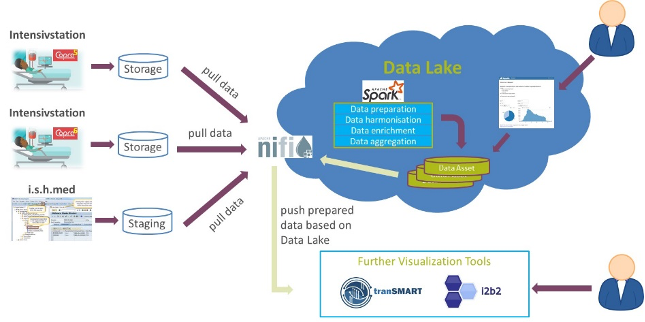
Abbildung 1: Konzeptionelle Architektur zur Visualisierung von intensivmedizinischen Daten zu COVID-19
Vollumfängliche Analyse durch Data Value Workshops
Um diese Aufgabe zu lösen, mussten zunächst die benötigen Datenquellen und Datenobjekte identifiziert werden. Durch die gute Zusammenarbeit mit den BIH- und Charité-Teams konnten relevante Teildatenbereiche der Patientendatenmanagementsysteme (PDMS) COPRA5 und COPRA6 sowie weitere Teildatenbereiche des Krankenhausinformationssystems (KIS) schnell als Datenquellen für diesen Use Case identifiziert werden. Als Datenobjekte wurden Patientenstammdaten, Bewegungsdaten der Patienten und verschiedene Vital- und Laborwerte identifiziert.
Diese Informationen erlauben den Data Engineers des Implementierungsteams die Daten mit Hilfe des Plattform-Werkzeugs Apache Nifi in den Data Lake laden. Beim Laden der Daten wurde bereits darauf geachtet, dass diese datenschutzkonform nur pseudonymisiert und unter Berücksichtigung des Berechtigungskonzeptes der Plattform gespeichert werden.
Bedeutung der Datenqualität
Im Anschluss wurden die Daten aufbereitet, harmonisiert, angereichert und schließlich auch aggregiert. Die Transformationen werden dabei mit Hilfe von Apache Spark und mehreren Data Pipelines umgesetzt. Die besondere Herausforderung hierbei bestand darin, die unterschiedlichen Datenmodelle der Quellsysteme zu integrieren, um sogenannte Data Assets zu erstellen, die als Basis für die spätere Visualisierung der Daten dienten.
Nach der Bereitstellung der Daten in Form von Data Assets begannen die Data Analysts und Data Scientists des Implementierungsteams mit ihrer Arbeit um die Daten für die Fachabteilung visuell darzustellen und auszuarbeiten. Um die Bearbeitungen kollaborativ zu entwickeln, wurde das Werkzeug Apache Zeppelin verwendet. Mit diesem Tool waren die Kollegen in der Lage, auf die bereitgestellten Data Assets direkt und ohne Umwege zuzugreifen, sowie mit Hilfe von Apache Spark ebenso wie Python und Bokeh diese interaktiv zu visualisieren.
Neben den neuen und modernen Werkzeugen existieren im medizinischen Forschungsumfeld auch etablierte Systeme zur Auswertung und Visualisierung von Daten, wie tranSMART oder i2b2. Um zu zeigen, dass der Data Lake auch als Datenlieferant für solche Systeme dienen kann, wurden zudem qualitativ hochwertige Data Assets aus dem Data Lake wieder an die benannten Systeme ausgeleitet und dort ebenfalls visualisiert. Der Data Lake konnte somit erfolgreich zeigen, dass er durch offene Standards auch problemlos mit Umsystemen interagieren kann.
Ergebnis
Mit Hilfe des Data Lakes und Experten von SVA, BIH und Charité ist es nun möglich innerhalb weniger Tage im Kontext der SARS-CoV2-Pandemie, Daten aus operativen Quellsystemen in den Data Lake zu importieren. Diese werden dort zu qualitativ hochwertigen Data Assets aufbereitet und anschließend für die Beantwortung von medizinischen Fragestellungen unterstützend verwendet.
Die nachfolgende Abbildung zeigt ein Ergebnis der Visualisierungen:
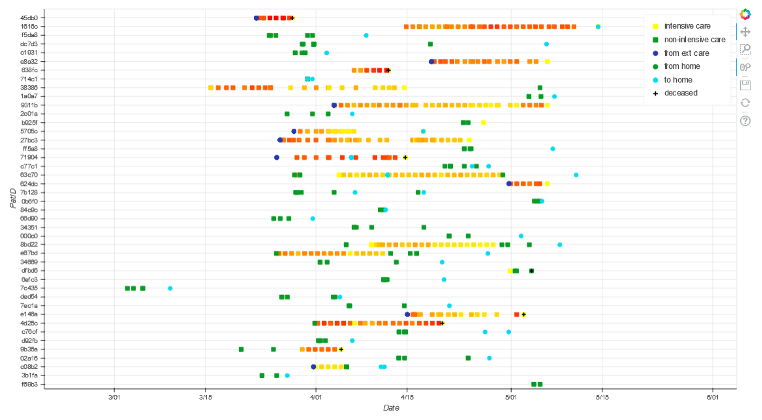
Abbildung 2: Visualisierung der Krankheitsverläufe und Bewegungen von Intensivpatienten mit COVID-19 (zeitlicher Ausschnitt)
Die Darstellung zeigt einen zeitlichen Ausschnitt der Entwicklung der Krankheitsverläufe sowie deren Endpunkte einzelner Patienten mit COVID-19, die durch die Intensivmedizin betreut wurden. Die Krankheitsverläufe sind dabei im Farbband von gelb bis rot gekennzeichnet. Gelb steht für einen niedrigen SOFA-Score und Rot für einen hohen SOFA-Score. Zudem werden ausgewählte Bewegungen der Patienten dargestellt.
 Marcel Fisser
Marcel Fisser